Microscopie Imagerie Cytométrie Azur
La plateforme MICA développe des solutions instrumentales et méthodologiques pour donner accès à de nouvelles modalités d'imagerie des échantillons biologiques.
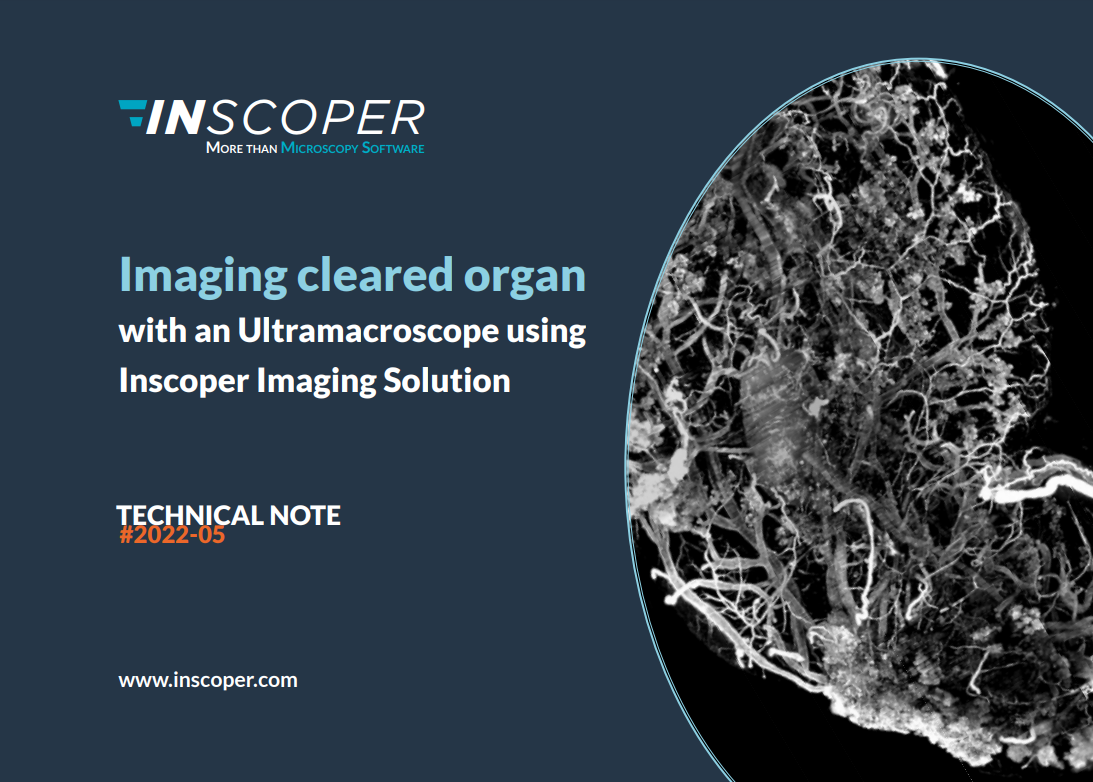
Ultramacroscope 

Ce système est un prototype original, disponible sur la plateforme pour imager vos échantillons et peut être dupliqué sur demande. Il a été développé en collaboration avec le Service de Mécanique Mutualisé (S2M) de l'Observatoire de la côte d'azur avec le soutien du GIS IBiSA et initié dans le contexte d'Actions Nationales de formation CNRS du Réseau Technologique de Microscopie de Fluorescence Multi-dimensionnelle.
- Principe et finalité :
-
Les coupes histologiques fines ne permettent pas de transmettre une information spatiale globale sauf au prix de reconstructions et recalages laborieux d'images. L’« Optical Coherence Tomoraphy » et l’« Optical Projection Tomography » (Sharpe et al., 2002) ont été spécifiquement développées pour de grands échantillons (1mm à 1 cm) pour obtenir des reconstitutions tridimensionnelles d’objets à partir de leurs projections selon différent angles. La résolution de ces techniques est de l’ordre de quelques microns. La microscopie confocale et bi-photons sont limitées en profondeur (300 à 700µm), et la résolution axiale de ces microscopies (z) est inférieure à la résolution latérale. Au début des années 1990, le développement du microscope confocal thêta (Linde et Stelzer, 1994). a fourni un nouvel outil pour l'étude des échantillons avec une résolution 3D élevée et isotrope. Le principe fondamental est la détection de la lumière de fluorescence à un angle de 90 degrés par rapport à l'axe d'éclairage. Par ce biais une nouvelle approche du sectionnement optique a été envisagée en illuminant le spécimen avec une feuille de lumière dans un plan perpendiculaire à la détection. L'équipement d'éclairage à feuille de lumière développé sur la plateforme est basé sur le principe de l'ultramicroscope (Dodt, et al., 2007). Il permet d'imager en 3 dimensions des échantillons biologiques transparents ou clarifiés sans nécessité d'effectuer des coupes sériées. Il a été pensé pour être modulaire et ouvert sur les dispositifs de détection disponibles dans le commerce.
- Caractéristiques :
-
- Le système peut imager des objets de tailles comprises entre 1-3 millimètres jusqu'à 1-3 centimètres.
- Il possède un double éclairage de l'échantillon pour obtenir des images homogènes d'échantillons transparents de grande taille.
- Il peut se positionner sous tous les macroscopes commerciaux (MVX10 Olympus, AF100 Nikon, Leica Z16 APO, Axio Zoom.V16 Carl Zeiss)
- Détails techniques des systèmes disponibles :
-
- Source de lumière : banc laser (Oxxius LBX-4C) avec 2 sorties fibrées monomode commutables 0/100% d'intensité. 4 lasers (P>40mW en sortie de fibre) : 405, 488, 561, 640 nm
- Optique de mise en forme des faisceaux : doublet achromatique suivi d'un diaphragme puis d'une lentille cylindrique de focale 10 cm : résolution axiale du système à 488nm = 5µm
- Porte échantillon : l'échantillon est translaté verticalement et perpendiculairement aux faisceaux qui le traversent dans une cuvette spécialement conçue. L'optique et le porte échantillon sont solidarisés sur le banc qui constitue le module.
- Détection :Macroscope MVX10 Olympus équipé avec des objectifs Plan Apochromatiques 1X/0,25 et 2X/0,5 : plages de grandissement de 0,63X à 12,5X
- Roue de filtres 4 positions (Bleu 447/60, Vert 520/35, Rouge 612/69, Rouge lointain 679/41)
- Caméra sCMOS (Orca Flash4 Hamamatsu)
- Pilotage du système avec le logiciel Inscoper
- Source de lumière : banc laser (Oxxius LBX-4C) avec 2 sorties fibrées monomode commutables 0/100% d'intensité. 4 lasers : 405, 488, 561, 640 nm
- Optique de mise en forme des faisceaux : doublet achromatique suivi d'un diaphragme puis d'une lentille cylindrique de focale 10 cm : résolution axiale du système à 488nm = 5µm
- Porte échantillon : l'échantillon est translaté verticalement et perpendiculairement aux faisceaux qui le traversent dans une cuvette spécialement conçue. L'optique et le porte échantillon sont solidarisés sur le banc qui constitue le module.
- Détection : Macroscope Z16 APOA Leica
- Roue de filtres 4 positions (Bleu 447/60, Vert 520/35, Rouge 612/69, Rouge lointain 679/41)
- Caméra Zyla (Andor)
- Pilotage du système avec le logiciel µManager
- Posters, Notes techniques & Publications :
-
Système Hilo/TIRF 
Ce système est un prototype de la famille des microscopes "super-résolution". Il atteint une résolution supérieure à 30nm en 3 couleurs avec des marquages classiques et est également compatible avec l'imagerie en vivant. Sa faiblesse est d'être dédié aux processus de culture cellulaire.
Il a été développé conjointement par l'équipe Morpheme et MICA, en collaboration avec l'équipe d'Ellen van Obberghen de l'iBV. Ce projet a été soutenu par des financements du GIS IBiSA et de l'Université Nice-Côte d'Azur.
- Principe et finalité :
-
Les approches de super-résolution ont été développées (STED, PALM/StORM, SIM) pour dépasser (ou plutôt "contourner") la limite de diffraction. Au fil des années, ces différentes techniques n'ont cessé de s'améliorer et présentent aujourd'hui l'énorme avantage d'offrir de très hautes performances tant en résolution latérale (jusqu'à 20 nm pour une image 2D) qu'en résolution axiale (jusqu'à 100 nm pour une image 3D). Malheureusement, lors de l'utilisation de ces techniques, d'énormes quantités d'énergie sont projetées sur l'échantillon et celui-ci doit souvent être marqué avec des marqueurs fluorescents très spécifiques, ce qui rend difficile son utilisation pour des échantillons vivants.
Pour certaines applications, il est plus crucial de pouvoir améliorer la résolution axiale et d'examiner simultanément plusieurs molécules en utilisant des molécules fluorescentes standard ou des protéines fluorescentes.
La méthode MA-TIRF (multiple-angle total internal reflection fluorescence) est particulièrement adaptée dans ce contexte. Initiée dans les années 90, cette technique utilise une des propriétés de la lumière, "l'onde évanescente". Lorsqu'un échantillon est éclairé par une source lumineuse très oblique, l'onde évanescente ne pénètre que de quelques centaines de nm (c'est le principe standard du TIRF). En jouant sur l'angle d'illumination, on peut faire varier la pénétration. Et en combinant précisément plusieurs angles d'illumination, on peut obtenir des informations supplémentaires. Mais cela nécessite un algorithme de reconstruction très spécifique. - Caractéristiques :
-
- Le microscope peut acquérir jusqu'à 3 couleurs de séquentiellement
- La résolution axiale est supérieure à 30nm
- L'épaisseur maximale est d'environ 500 nm (pour la reconstruction)
- La résolution temporelle est d'environ 1s par marquage pour obtenir une image 3D
- La position axiale est absolue par rapport à la surface de la lamelle
- Détails techniques du système disponible :
-
- Source de lumière : 488nm, 561nm, 640nm. 405nm est également disponible mais pas pour la reconstruction 3D
- Les échantillons doivent être dans un milieu de culture ou du PBS sur une lamelle #1.5 (170µm d'épaisseur) sur une lame ou une boîte de Pétri de 35mm
- Détection : le microscope est un Nikon Ti-E équipé avec un objectif 100x/1.49O
- Caméra EM-CCD Andor Ultra 2
- Pilotage du système avec un programme Labview fait maison
- L'analyse est effectuée en ligne par un algorithme Matlab fait maison avec une interface conviviale dédiée
- Applications :
-
- Matrice extra-cellulaire, adhésion cellulaire
- Endocytose, exocytose
- Trafic cellulaire
- Compatible avec l'imagerie sur cellules vivantes (projet en cours)
- Posters & Publications :
-
- Nanometric axial resolution of fibronectin assembly units achieved with an efficient reconstruction approach for multi-angle-TIRF microscopy. Soubies E, Radwanska A, Grall D, Blanc-Féraud L, Van Obberghen-Schilling E, Schaub S. Sci Rep. 2019 Feb 13;9(1):1926. doi: 10.1038/s41598-018-36119-3.
- Improving 3D MA-TIRF Reconstruction with Deconvolution and Background Estimation. Emmanuel Soubies, Laure Blanc-Féraud, Sébastien Schaub, Ellen Van Obberghen-Schilling. ISBI 2019 - IEEE International Symposium on Biomedical Imaging, Apr 2019
- A Framework for Multi-angle Tirf Microscope Calibration. Emmanuel Soubies, Sébastien Schaub, Agata Radwanska, Ellen Van Obberghen-Schilling, Laure Blanc-Féraud. ISBI - International Symposium on Biomedical Imaging, Apr 2016
- The Valrose Biology Institute is now equipped with an optical microscope with molecular resolution. Université Côte d’Azur News
- Un microscope optique à résolution moléculaire. CNRS - INSB News